2021年11月1日,浙江大学任艾明课题组与华东理工大学杨弋课题组合作在Nature Chemical Biology杂志上发表题为“Structure-based investigation of fluorogenic Pepper aptamer”的研究论文,报道了新型RNA荧光适配体Pepper与染料配体HBC以及其六种类似物的晶体结构,揭示了不含G-四联体的Pepper适配体识别染料配体的机制。
RNA分子在生物体内具有重要的生物学功能且其具备十分复杂的动态变化。活细胞RNA成像技术可对RNA分子进行实时监测,用以研究其转录、翻译、转运、剪切、定位、降解等动态过程。荧光RNA适配体是近些年来发展的一种简单而有效的RNA分子荧光标记工具,其可结合并固定在溶液中不发荧光的染料小分子,进而发出强烈的荧光,目前已被广泛应用于RNA活细胞成像。
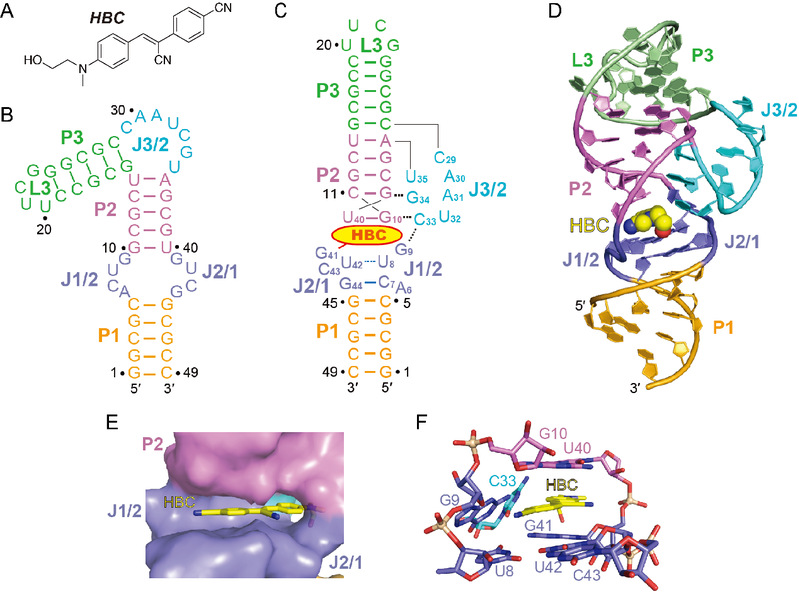
HBC((4-((2-羟乙基)(甲基)氨基)-亚苄基)-氰基苯乙腈)是基于绿色荧光蛋白GFP的荧光团HBI(4-羟基亚苄基咪唑啉酮)开发的一种新型染料配体(图1A)。通用SELEX(指数富集的配体系统进化)技术体外筛选得到的Pepper适配体可以特异地结合HBC并激活其荧光,其二级结构如图1B所示。为了阐明Pepper适配体识别配体HBC的机制,任艾明课题组采用X-射线晶体学的方法解析了Pepper-HBC复合物的晶体结构,与绝大多数已有的RNA荧光适配体不同,其三级结构中不包含G-四联体。
Pepper-HBC复合物的整体结构呈音叉状,包括三个双螺旋P1、P2和P3,并由三个Junction区域J1/2、J2/1和J3/2连接,(图1-C, D)。J3/2和J1/2以及P2形成远距离相互作用,帮助维持整体三维结构的稳定(图1C)。配体HBC结合于整体结构的中心位置(图1D),以近平面构象堆积于非G-四联体四碱基平面(U8, G41, U42, G43)和G10-U40非经典碱基对之间(图1E),并由碱基对G9-C33从侧面固定(图1-D, E)。另外,复合物结构中存在若干镁离子,对维持整体结构的稳定以及配体结合口袋的形成至关重要。体外荧光实验结果揭示了不同离子对Pepper适配体结合并激活HBC荧光的影响,也验证了关键核苷酸对Pepper适配体结合染料配体HBC的重要性。另外,本文也解析了Pepper适配体与六种HBC类似物的复合物晶体结构,其整体结构以及配体结合口袋均与Pepper-HBC复合物类似,深入揭示了Pepper适配体与配体之间的结合机制。
本项研究为进一步优化Pepper适配体的序列以及设计具备更佳光化学特性的染料分子提供了结构基础,也推动了其在活细胞RNA成像技术中的应用。在RNA分子的三维结构探讨上,本研究进一步补充和丰富了目前相对有限的RNA三维结构数据库。
该项研究工作得到了国家自然科学基金(32022039,31870810,91940302,91640104),浙江省杰出青年基金(LR19C050003)以及浙江大学启动基金的资助。浙江大学生命科学研究院已毕业博士研究生黄凯怡以及华东理工大学的陈显军副研究员为本文的共同第一作者。
附:本实验室长期专注于RNA分子(核开关、核酶、核酸适配体等)的高级结构研究,并基于结构开发RNA分子的相关应用。欢迎感兴趣的具有结构生物学、生物化学、分子生物学、化学合成相关研究背景的研究人员申请本实验室博士后或研究助理岗位(详情见/25710/list.htm)。
原文链接:https://www.nature.com/articles/s41589-021-00884-6




